I have a vcf file. I need to update the ID column in my vcf file. This is how my vcf file look like: 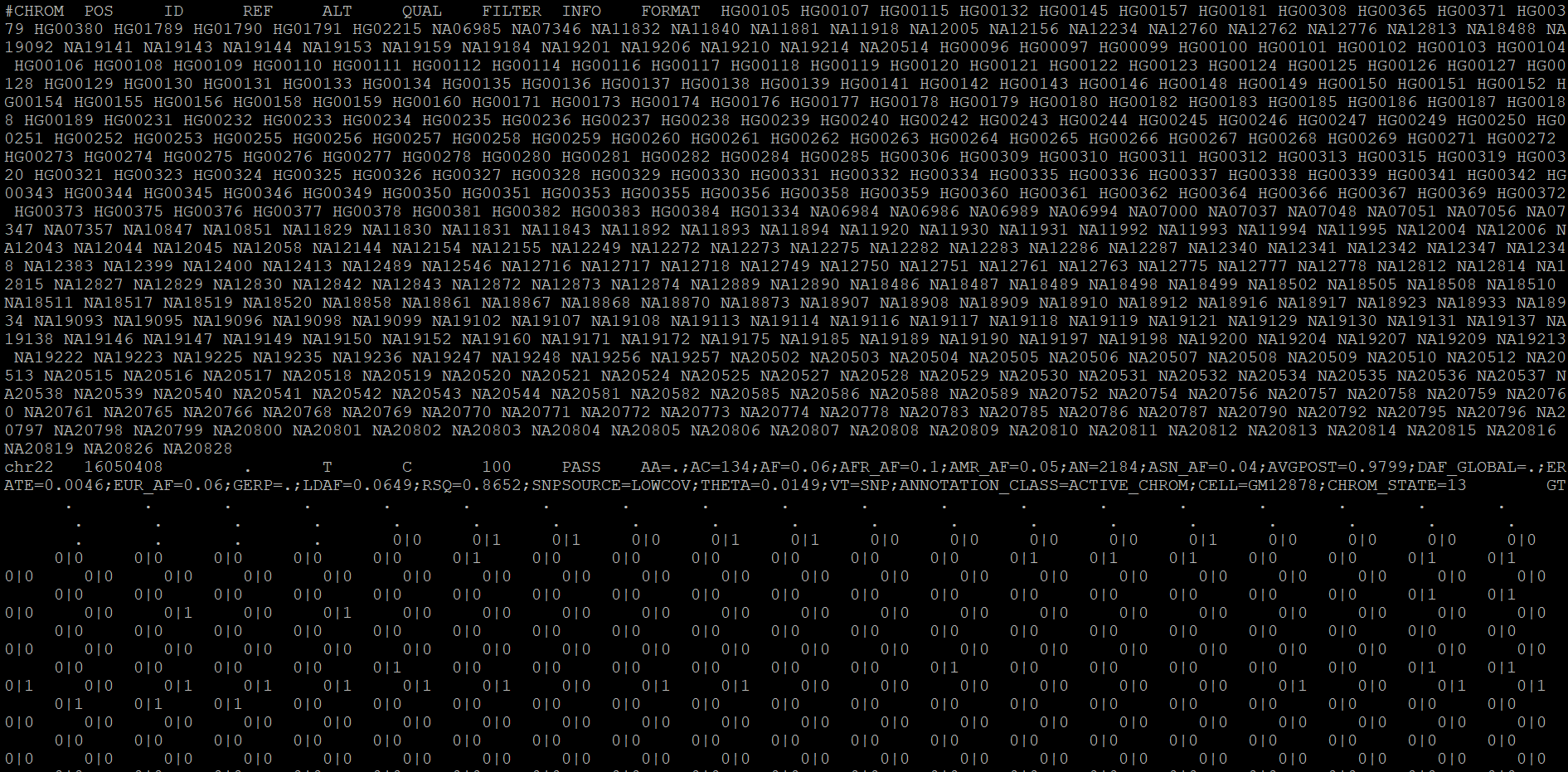
0797 NA20798 NA20799 NA20800 NA20801 NA20802 NA20803 NA20804 NA20805 NA20806 NA20807 NA20808 NA20809 NA20810 NA20811 NA20812 NA20813 NA20814 NA20815 NA20816 NA20819 NA20826 NA20828
chr22 16050408 . T C 100 PASS AA=.;AC=134;AF=0.06;AFR_AF=0.1;AMR_AF=0.05;AN=2184;ASN_AF=0.04;AVGPOST=0.9799;DAF_GLOBAL=.;ERATE=0.0046;EUR_AF=0.06;GERP=.;LDAF=0.0649;RSQ=0.8652;SNPSOURCE=LOWCOV;THETA=0.0149;VT=SNP;ANNOTATION_CLASS=ACTIVE_CHROM;CELL=GM12878;CHROM_STATE=13 GT . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 0|0 0|1 0|1 0|0 0|1 0|1 0|0 0|0 0|0 0|0 0|1 0|0 0|0 0|0 0|0 0|0 0|0 0|0 0|0 0|0 0|1 0|0 0|0 0|0 0|0 0|0 0|0 0|1 0|1 0|1 0|0 0|0 0|1 0|1 0|0 0|0 0|0 0|0 0|0 0|0 0|0 0|0 0|0 0|0 0|0 0|0 0|0 0|0 0|0 0|0 0|0 0|0 0|0 0|0 0|0 0|0 0|0 0|0 0|0 0|0 0|0 0|0 0|0 0|0 0|0 0|0 0|0 0|0 0|0 0|0 0|0 0|1 0|1 0|0 0|0 0|1 0|0 0|1 0|0 0|0 0|0 0|0 0|0 0|0 0|0 0|0 0|0 0|0 0|0 0|0 0|0 0|0 0|0 0|0 0|0 0|0 0|0 0|0 0|0 0|0 0|0 0|0 0|0 0|0 0|0 0|0 0|0 0|0 0|0 0|0 0|0 0|0 0|0 0|0 0|0 0|0 0|0 0|0 0|0 0|0 0|0 0|0 0|0 0|0 0|0 0|0 0|0 0|0 0|0 0|0 0|0 0|0 0|0 0|0 0|0 0|0 0|1 0|0 0|0 0|0 0|0 0|0 0|0 0|1 0|0 0|0 0|0 0|0 0|0 0|1 0|1 0|1 0|0 0|1 0|1 0|1 0|1 0|1 0|0 0|1 0|1 0|0 0|0 0|0 0|0 0|0 0|0 0|1 0|0 0|1 0|1 0|1 0|1 0|1 0|0 0|0 0|0 0|0 0|0 0|0
Now I want to update the ID column which is represented as "." by chr22_16050408_T_C_b37 so each ID column in my vcf file should look like chr{no.}_position_refallele_altallele_b37.
So, earlier the ID column is .
Now I want to update it with:
chrr22_16050408 _T_C_b37
I tried to use the following command but its now giving me the answer.
awk 'NR>1 {print $1""$2""$3""$4"_b37"}' file1.txt